RT-PCR = 逆转录 + PCR 或 qPCR(Real-time PCR)
- RT-PCR:逆转录 PCR
- qPCR:定量(quantitative)PCR
- Real-time PCR:即时 PCR
逆转录
- 逆转录 | 反转录
- 在逆转录酶和引物的作用下,以样本中提取的 RNA 为模板,合成出 cDNA(complementary DNA,互补 DNA)。
逆转录酶
- RNA 指导的 DNA 聚合酶活性(RNA → DNA):RNA 指导的 DNA 聚合,逆转录的关键;
- RNase H(Hybrid)活性:水解杂化双链中的 RNA 单链,还会和聚合酶活性竞争样品中的 RNA 与引物或 cDNA 延伸链形成的杂合双联;
- DNA 指导的 DNA 聚合酶活性(DNA → DNA):DNA 模板也可直接用于/参与逆转录,所以要用 DNase 去除样品中的 DNA。
在选择逆转录酶时,建议选择无 RNaseH 活性(RNaseH-)的逆转录酶。具有 RNaseH 活性的逆转录酶的 RNaseH 活性会与聚合酶活性竞争 RNA 模板与 DNA 引物(或 cDNA 延伸链)形成的杂合链,并降解杂合链中的 RNA 链。被 RNaseH 活性所降解的 RNA 模板不能再作为合成 cDNA 的有效底物,降低了 cDNA 合成的产量与长度。
引物
用于反转录的引物有随机引物、通用引物(Oligo-dT)及基因特异性引物三种,下表为三种引物的介绍:
常见的三种引物
| 引物 | 适用范围 |
|---|---|
| 随机引物 | 适用于长的或具有发卡结构的 RNA。适用于 rRNA、mRNA、tRNA 等所有 RNA 的反转录 反应。主要用于单一模板的 RT-PCR 反应 |
| Oligo-dT | 适用于具有 PolyA 尾巴的 RNA(原核生物的 RNA、真核生物的 rRNA 和 tRNA 不具有 PolyA 尾巴) |
| 基因特异性引物 | 与模板序列互补的引物,适用于目的序列已知的情况 |
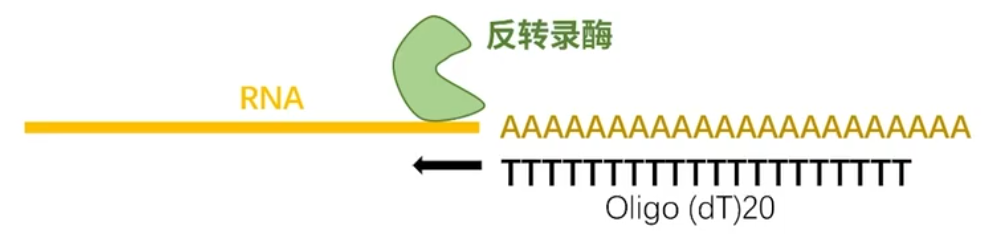
Oligo-dT
- Oligo-dT 需要 RNA 有 PolyA 尾,即需要 RNA 结构完整。
qPCRReal-time PCR
即时聚合酶链式反应是一种在 DNA 扩增反应中,以荧光染剂侦测每次聚合酶链锁反应(PCR)循环后产物总量的方法。
- “荧光染剂”:如 SYBR Green,其在结合双链 DNA 时会发出荧光。PCR 扩增过程中,随着 DNA 的合成,SYBR Green 与新合成的双链 DNA 结合,从而增加荧光信号的强度。
- “Real-time”:在每个 PCR 循环结束时,荧光信号的强度被实时测量。通过监测荧光信号的变化,可以在反应的早期阶段判断 PCR 扩增的进程。
- “q(quantitative)”:通过与标准曲线进行比较,SYBR Green 可以帮助定量样本中目标 DNA 的初始浓度。荧光信号的强度与 DNA 的量成正比。
qPCR 结果评价指标:
- CT(cycle threshold):指扩增过程中,荧光信号首次达到设定阈值的循环数。换句话说,就是检测到显著荧光信号所需的 PCR 循环次数。
- CT 值与初始模板浓度成反比。
- CT 值 SD 应 < 0.05。
- 扩增曲线(amplification plot):指PCR过程中,以循环数为横坐标,以反应过程中实时荧光强度为纵坐标所做的曲线。理想的扩增曲线应呈现出光滑的 S 型曲线(扩增曲线 log 图则不然):
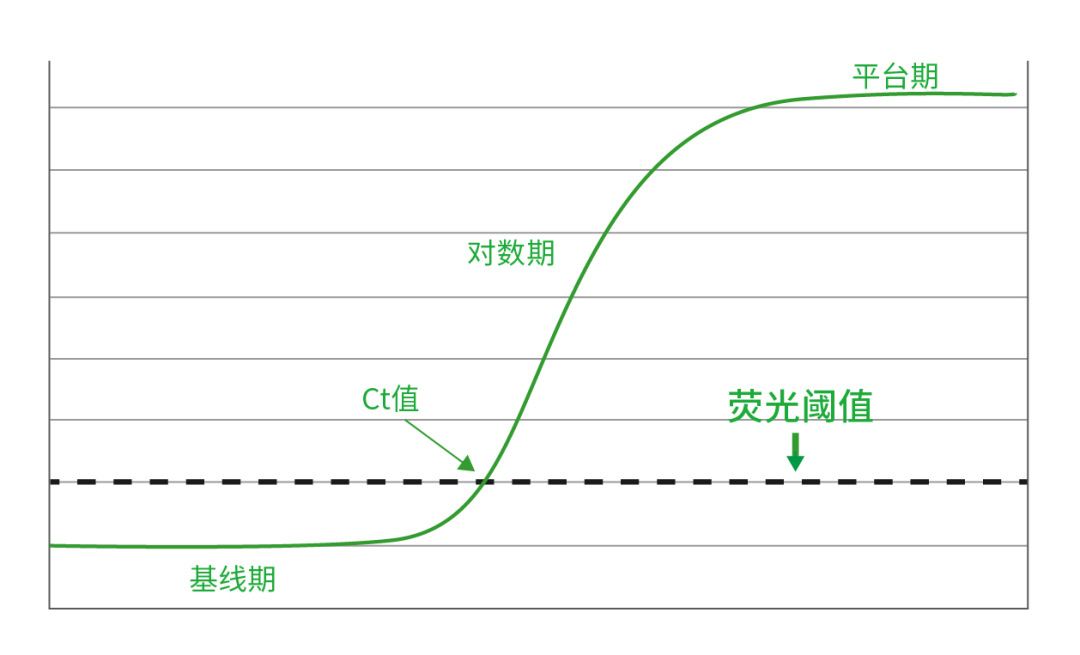
扩增曲线示意图
- 熔解曲线(求导曲线):在循环扩增结束后,再设置一段由65℃逐渐升温到95℃(具体温度可能要视情况而定)的程序。随着 DNA 解链变性,荧光染剂从 DNA 中释放,导致荧光信号降低。
- 普通的溶解曲线是逐渐下降的 S 形曲线,Y 轴是荧光信号强度。
- 通常所说的溶解曲线是指普通溶解曲线的负一阶导数曲线,Y 轴是指熔解曲线的荧光信号强度随温度变化的斜率的负值。这个曲线中的峰对应的温度即为解链温度 Tm——一半 DNA 变性时的温度。

溶解曲线
qPCR 数据分析:
https://www.bilibili.com/video/BV1eU4y1o7uR
$$\Delta CT_{目的} = CT_{目的}-Mean(CT_{内参}) \,.$$$$\Delta \Delta CT_{实验组目的} = \Delta CT_{实验组目的}-Mean(\Delta CT_{对照组目的}) \,.$$最后得到目的基因相对表达量:
$$RelExpr = 2^{-\Delta \Delta CT_{实验组目的}} \,.$$另一种方法:
$$X = 2^{-(CT_{目的}-CT_{内参})} \,.$$$$Y = X - Mean(X) \,.$$应该满足:各组的 $Mean(Y)=1$。
各实验组计算相对表达量:
$$RelExpr = Mean(X - Mean(X_{对照})) \,.$$梯度 PCR
琼脂糖凝胶电泳
琼脂糖
- 琼脂糖是琼脂的一种成分,它形成了一个由螺旋状的琼脂糖分子组成的三维凝胶基质,这些螺旋状的琼脂糖分子在超线圈束中被氢键固定,有通道和孔隙,分子能够通过。当加热时,这些氢键断裂,使琼脂糖变成液体,并允许它在复位前被倒入模具。
- 琼脂糖在凝胶中的百分比影响了孔的大小,从而影响了可能通过的分子的大小以及通过的速度。琼脂糖的百分比越高,孔径越小,因此能够通过的分子越小,迁移速度越慢。
- 在分子生物学实验室中,0.7–1% 的琼脂糖凝胶通常用于日常的 DNA 分离,对 0.2–10kb 范围内的片段提供良好、清晰的区分。较大的片段可以用较低百分比的凝胶来解决,但它们会变得非常脆弱且难以处理,而较高百分比的凝胶会对小片段提供更好的分辨率,但很脆且可能凝固不均匀。
溴化乙锭
- 由于 DNA 在肉眼中是不可见的,在凝固过程中,会向凝胶中加溴化乙锭(EthBr)。EthBr 是一种嵌入剂,当暴露在紫外线下时,它会发出橙色萤光,与 DNA 结合后强度增强近 20 倍。
步骤
- 1.2g 琼脂糖粉末,80ml TAM(用 dd 水配成 1x),混匀,用锡纸封口,微波炉加热 2min
- 稍冷却(注意不要凝固了)之后加入 8ul(1/10000)SYBR 显影剂
- 胶板与模具板装好,倒入胶液,两头胶板与模具板之间见到溢出提示量足够
- 冷却后去除胶板,放入电泳池,加 TAM 至没过胶面 5mm 左右
- 计算:之前 qPCR 的 96 孔板,每一组(比如 造模后 0h 的 内参组)选择比较一致的至少 3 个孔,数量为 N。
- 准备一张封口胶,在上面加 loading buffer 1ul x N(注意第二挡不要按到底,以免产生气泡影响后续操作),每滴加 cDNA 溶液(“计算”中选择的孔)10ul 混匀,加入上样孔
- 开始电泳,约 120V 35min(电压过高可能导致凝胶熔化)。
- 使用紫外光显影 DNA 条带(溴化乙锭 EthBr)(注意防护)(或者直接观察 SYBR 荧光?)。